【Chainer】Chainerを利用したスネオトンガリ分類
Chainerを使ってみたかったので何かしら画像分類をしようと思う.
あだち充の大ファンであるのであだち分類しようと思ったがさすがに人間に不可能なことは
コンピュータにもまだ無理だろうと考えてスネオとトンガリくらいだったらいけると踏んだ.
Google画像検索からトンガリのサンプル,スネオのサンプルを収集する.

スネオはまあまあ集まるがトンガリのサンプルが全くもって少なすぎる.
Data augmentationを施行して水増しした.
PFN発のディープラーニングフレームワークchainerで画像分類をするよ(chainerでニューラルネット1) - 人工言語処理入門
こちらを参考にさせて頂いた.まずは平均画像mean.npyを作成.
python /***/***/examples/imagenet/compute_mean.py train.txt
後は
python /.../examples/imagenet/train_imagenet.py -g -1 /.../train.txt /.../test.txt 2>&1
で分類が始まる.caffeの導入に比べて非常に簡単に事が進んだ.
が、3分程でMemoryErrorで強制終了と相成った.スペックの不足と考えられる.
batchsizeを少なくすると動くようであるが見送った.
【論文】Spindle Cell Melanoma vs Desmoplastic Cell Melanoma (Pathology Informatics)
Spindle Cell MelanomaとDesmoplastic Cell Melanomaの分類論文を読んだ。 nuclear circularityの違いにより診断ができるかということをどちらもn=9でImageJを使った閾値の検出で分類を行った論文.ウルム大学(アルバート・アインシュタインの出生の地)のペーパーである.
こちらで全文が読むことができる.
Figure2でImage-Jのthreshold-based segmentationにより核を同定,そこからedge detectionを行い形態をCircularity,Aspect Ratio,Solidityで評価している.
この方法で例えば20%未満のspindled nucleiもしくは20%以上のround nucleiがあると判断されればその症例はCSMとされる.
この分類での全体の正答率は89%であったとのこと.
【Python】Pythonで京大数学を解く 2003前
京大数学をパソコンで解こうという試み.
matplotlib 3Dの勉強をしたいという気持ちからはじめました.
参考にしたのはこちらです.とても勉強になります.
http://mathesaurus.sourceforge.net/r-numpy.html
まずは京大で良く出るといわれている回転体の体積.
曲線をX軸の周りに回転して得られる体積~というヤツである.
これを3Dでプロットしようと思います.
京大03前
f(x) = xsinx (x>=0)とする.点π/2,π/2におけるy=f(x)の法線とy=f(x)のグラフの0<=x<=π/2の部分,及びy軸とで囲まれる図形を考える.この図形をx軸の周りに回転して得られる回転体の体積を求めよ.
Numpyで解いてみようと思っていたので,挑戦.
法線はy = -x+πであることからとりあえず2Dに図示.


これらをY-axisの周りに一回転すればいいわけだから,

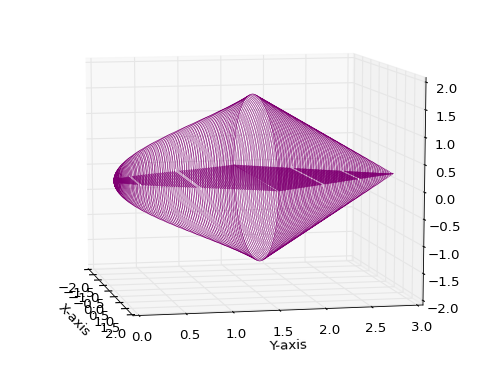
体積はまた今度求めようと思います.